孕激素受體通過RUNX轉錄因子相互作用和染色質重塑介導排卵轉錄
孕激素受體(PGR)在生殖組織中扮演多種角色,從而調控哺乳動物的生育能力。在卵巢中,通過對一組獨特基因的轉錄調控,迅速、急性誘導PGR是排卵的關鍵決定因素, 最終導致卵泡破裂。然而,關于PGR在排卵中的專門功能的分子機制尚不清楚。我們通過組合ATAC-seq、RNA-seq和ChIP-seq分析,在野生型和特定異構體PGR敲除小鼠中,建立了詳細的PGR作用基因組譜系。我們證明,刺激排卵迅速重塑了三分之二的染色質可及性,與基因表達的改變相關。我們觀察到,卵巢特異性的PGR作用與RUNX轉錄因子的相互作用有關,70%的PGR結合區域也與RUNX1結合。這些轉錄復合物將PGR結合到近端啟動子區域。此外,PGR與典型的NR3C基序的直接結合使染色質具有可及性。所有這些PGR作用共同介導了必需的排卵基因的誘導。我們的發現突出了一種新的針對排卵的PGR轉錄機制,為不孕不育治療或阻止排卵的新避孕藥提供了新的靶點。
圖形摘要

該研究于2023年4月發表發表在《Nucleic Acids Research》,IF:14.9。
技術路線
結果
1、排卵刺激通過不同的轉錄因子結合重塑染色質狀態和轉錄
通過hCG注射小鼠誘導排卵提供了一個體內模型,用于研究顆粒細胞對排卵信號的快速反應。比較ATAC-seq揭示了染色質可及性和排卵轉錄因子結合的動態變化。嚴格的ATAC-seq峰值調用標準確定了總共71,287個可訪問的染色質峰,其中三分之二的峰在黃體生成素(LH)刺激后的6小時內發生了改變,有28,729個位點(40.3%)受LH誘導,21,257個位點(29.8%)顯著抑制,21,301個位點(29.8%)可及性無變化(FDR ≤ 0.05,圖1A)。LH介導的可及性變化不僅發生在啟動子區域,還發生在遠端增強子和基因區域(圖1B)。
為了預測參與LH介導的開放染色質位點可及性變化的關鍵轉錄因子的活性,進行了轉錄因子結合模體的差異分析(圖1C)。許多已知的排卵轉錄因子(如JUN/FOS、RUNX、CREB和CEBP)的模體在LH刺激后開放染色質峰中的結合分數顯著增加,而CTCF、NR5A和ESRR的模體則富集于LH前開放染色質位點。有趣的是,NR3C家族的典型模體,包括孕激素受體(PGR)、糖皮質激素受體(NR3C1/GR)、醛固酮受體(NR3C2/MR)和雄激素受體(NR3C4/AR),在LH刺激前的開放染色質中顯示出顯著較高的結合分數。為支持這些發現,使用HOMER進行的新生模體富集分析也在LH誘導的開放染色質區域中鑒定出了JUN/FOS、RUNT和CEBP的模體,但沒有在LH抑制的開放染色質區域中發現(圖1D)。NR5A和GATA家族的模體在所有ATAC-seq峰集中同樣富集。重要的是,在LH抑制的子集中僅發現與NR3C成員結合的模體最匹配,與差異轉錄因子結合分析模式一致。轉錄因子直接結合到這些模體的證據可在聚合的足跡圖中觀察到(圖1E)。顯示與轉錄因子結合到這些位點相關的足跡模式,可以觀察到中度至深度的凹陷(JUN/FOS、CTCF、NR5A)和較淺的模式(RUNT、NR3C),暗示這些轉錄因子停留時間的差異。JUN/FOS足跡僅在LH刺激后才明顯,而NR3C足跡的證據在未刺激的細胞中最明顯。
通過對體內LH刺激前或LH刺激后8小時收集的顆粒細胞進行RNA-seq分析,將染色質可及性譜系與基因表達變化相關聯。共鑒定了2088個差異表達基因(DEG),其中52%受LH刺激上調(圖2A)。其中包括許多已知與排卵相關的基因,如Ptgs2、Pgr、Runx1、Runx2和Adamts1。使用Ingenuity Pathway Analysis(IPA)軟件預測LH DEG的潛在上游調控因子,確定了一系列轉錄因子,其中許多也是對LH激增作出反應的DEG,包括PGR(log FC = 6.4)、RUNX1(log FC = 4.6)、CEBPβ(log FC = 2.5)和JUNB(log FC = 2.1)。值得注意的是,除了PGR外,這些轉錄因子的結合模體也顯示出LH刺激后的可及性增加(圖1C、D)。通過比較LH DEG和這些基因啟動子(TSS上游3 kb內)的LH依賴性染色質可及性,展示了排卵前顆粒細胞中啟動子可及性與下游基因表達之間的全局關系。69.3%的LH驅動DEG(2088個基因中的1447個)在啟動子可及性發生顯著變化時顯示出轉錄變化(圖2B,紅色符號)。LH誘導的基因的啟動子可及性大多增加,而LH抑制的基因的可及性較低(Spearman相關系數=0.4297)。作為示例,Cxcr4和Inhbb是兩個分別上調和下調的基因,其表達模式與基因啟動子處的ATAC-seq信號明顯相關(圖2C)。然而,在某些情況下,差異調節的基因具有一直可及的啟動子,如Cited1。LH誘導的啟動子可及性高的轉錄活躍基因主要與排卵相關的生物過程相關,包括血管生成、MAPK信號通路和炎癥應答。總之,這些結果表明,作為對LH激增的全局染色質重塑,很可能通過激活一組特定的轉錄因子,驅動與LH觸發的排卵相關的基因表達譜系。
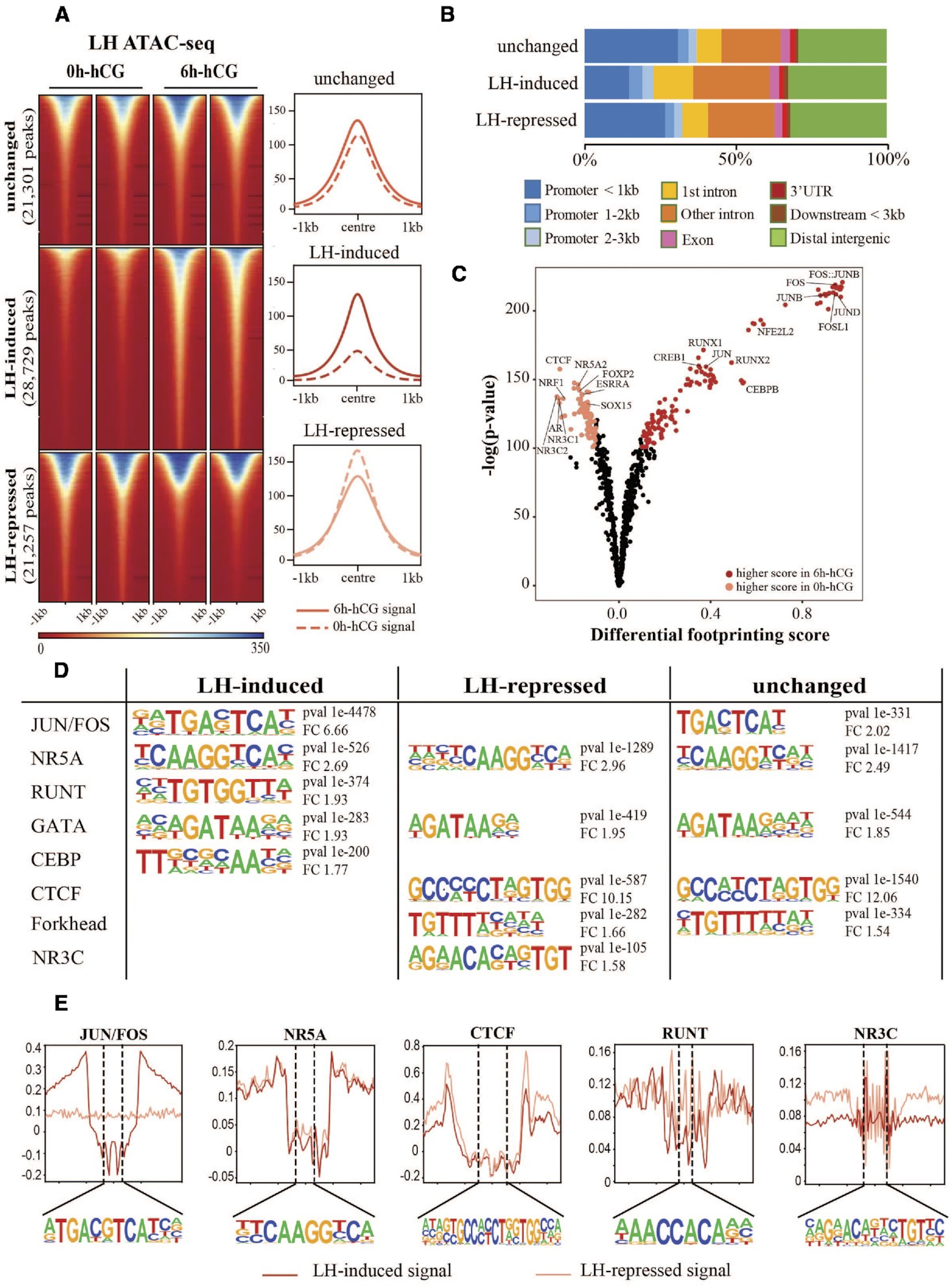
圖1.人絨毛膜促性腺激素治療后顆粒細胞染色質景觀的整體變化
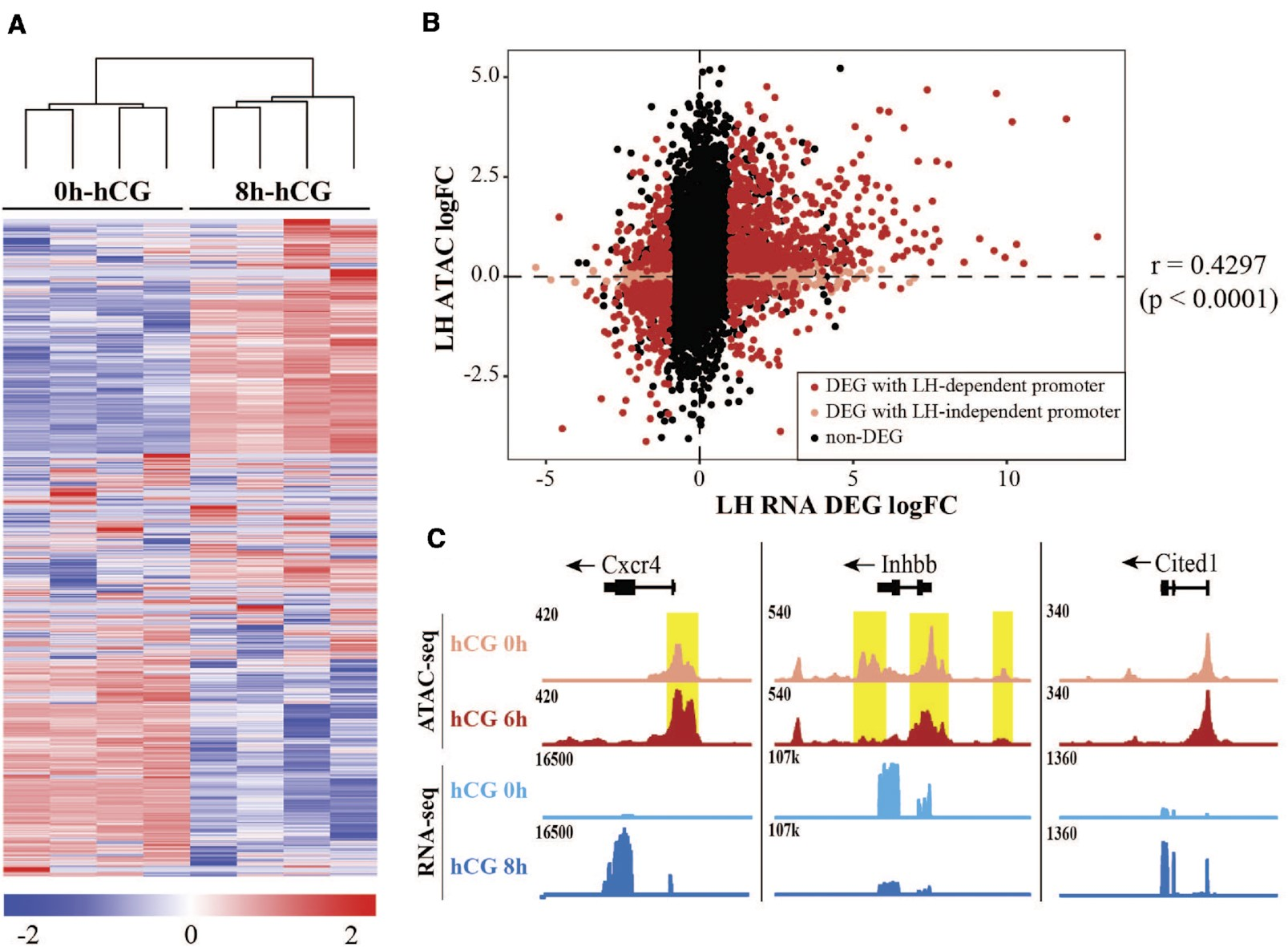
圖2.hCG治療后顆粒細胞的整體轉錄變化。
2、PGR和RUNX1在排卵期顆粒細胞中相互結合染色質
RUNX1在排卵中具有功能重要性,并且RUNT基序在圍絕經期顆粒細胞的PGR ChIP-seq峰值中高度富集。此外,我們發現在LH刺激后獲得染色質可及性的區域含有富集的RUNT基序(圖1C)。RUNX1和RUNX2的表達在顆粒細胞中對LH激素的刺激作出反應時被誘導,并且在顆粒細胞特異性Sf1-cre RUNX1 KO小鼠中排卵被延遲。為了確定RUNX1是否是介導排卵基因表達的轉錄調控機制的關鍵因素,我們使用ChIP-seq在體內hCG處理前或處理后的6小時內定義了顆粒細胞中RUNX1的全基因組靶點。在LH激素釋放前就檢測到全局RUNX1-染色質結合,但在LH激素釋放后也被7倍誘導(16065個LH誘導的峰值)(圖3A)。LH激活的RUNX1結合與LH誘導的開放染色質強烈相關(通過ATAC-seq定義)。通過比較RUNX1和PGR的ChIP-seq,我們在對LH刺激做出反應的顆粒細胞中確定了RUNX1和PGR之間的明顯關系。PGR和RUNX1在100 bp內共享了大量結合位點,其中9704個染色質位點(總PGR的70%或總RUNX1結合位點的52%)同時結合了PGR和RUNX1(圖3B)。在這些共享的PGR/RUNX1結合位點中,有9288個位于同一時間點鑒定的ATAC-seq開放染色質位點中。將轉錄活躍的RUNX1和PGR cistrome分為每個轉錄因子共享或only結合的部分,發現沒有RUNX1共結合的PGR結合位點與基因啟動子的關聯程度相對較低。在PGR/RUNX1共結合位點中,靠近啟動子的結合(在TSS的1 kb范圍內)是PGR的主要特點(圖3C)。PGR和RUNX1結合位點的顯著高度重疊,再加上我們先前報告的顆粒細胞PGR ChIP-seq中富集的RUNX1結合基序,暗示了PGR以先前報道的顆粒細胞特異性能力通過與RUNX1共結合來靶向近端啟動子。這種依賴關系不是相互的,因為RUNX1顯示出獨立于PGR共結合的近端啟動子占據的優勢。為了測試LH誘導的RUNX1染色質結合是否依賴于PGR,我們比較了在PGR重疊位點與獨立于PGR的位點上LH誘導的RUNX1結合強度。在RUNX1富集水平上沒有觀察到差異,這表明PGR不太可能在這些共享位點上發揮RUNX1的連接或招募作用。
通過將具有PGR和/或RUNX1結合的基因與我們的LH響應性差異表達基因列表進行比較,展示了PGR和RUNX1在基因表達中對排卵信號的重要性(圖3D)。值得注意的是,所有LH介導的差異表達基因中,至少62.4%的基因在其近端啟動子(在3 kb范圍內)上具有PGR或RUNX1結合,與LH抑制的差異表達基因相比,LH誘導的差異表達基因更頻繁地被PGR和/或RUNX1結合。其中,大多數(55%)RUNX1結合的LH差異表達基因和大多數(87%)PGR結合的LH差異表達基因實際上都被兩個轉錄因子共結合,這意味著PGR和RUNX1在目標啟動子上的同時相互作用是它們各自在基因調控中的主要機制。在排卵期顆粒細胞中的許多已知的PGR和RUNX1靶基因的啟動子中可以看到PGR/RUNX1的相互染色質占據(Rgcc,Adamts1和Cstl)(圖3E)。PGR/RUNX1的合作還可以通過形成與調控基因的近端啟動子接觸的遠端增強子來調控靶基因的表達。為了研究這一點,我們整合了公開可用的小鼠顆粒細胞Hi-C的染色質-染色質相互作用數據。Hi-C確定了與516/2088個LH差異表達基因的TSS相關的遠端染色質相互作用,其中至少28%被PGR和/或RUNX1結合(圖3D)。與PGR和RUNX1在近端啟動子上的結合不同,PGR/RUNX1在遠端增強子上的結合與LH誘導和LH抑制的基因表達同樣相關。以PGR靶基因Adamts1為例,它顯示了強烈的PGR/RUNX1共結合,位于通過小鼠顆粒細胞中的Hi-C以及人類卵巢中的啟動子捕獲-C指示的遠端區域與Adamts1 TSS相互作用(圖3F)。
基序富集分析顯示,期望的典型RUNT基序在RUNX1基因組中高度且顯著富集(相對于背景的3.5倍,P值為1e-787),以及PGR基因組中(相對于背景的2.8倍,P值為1e-370)(圖3G)。NR3C基序在RUNX1結合的基因組中也富集(相對于背景的2倍,P值為1e-25)。這種富集僅限于PGR/RUNX1共結合位點。其他富集的非典型基序包括與JUN/FOS、CEBP和GATA轉錄因子對應的基序。
通過近距離連接測定(PLA)證實了PGR和RUNX1在小鼠顆粒細胞核中的密切物理相互作用,這些細胞在模擬體內排卵刺激的情況下經過hCG和R5020處理長達8小時(圖4A)。PLA信號顯示LH刺激迅速短暫誘導了PGR/RUNX1復合物,表明在刺激前大部分時間不存在的PGR/RUNX1相互作用在刺激后4小時內增加。此外,通過免疫沉淀結合質譜法在PGR表達的人類顆粒細胞中,根據對R5020處理的響應,也表明了RUNX1和PGR之間的物理相互作用(圖4B)。PGR還可以與其他RUNX成員相互作用,正如在暴露于排卵刺激的顆粒細胞中的PGR/RUNX2 PLA所示(圖4C)。

圖3.在排卵周圍顆粒細胞中,PGR和RUNX1共有共同的染色質靶點。
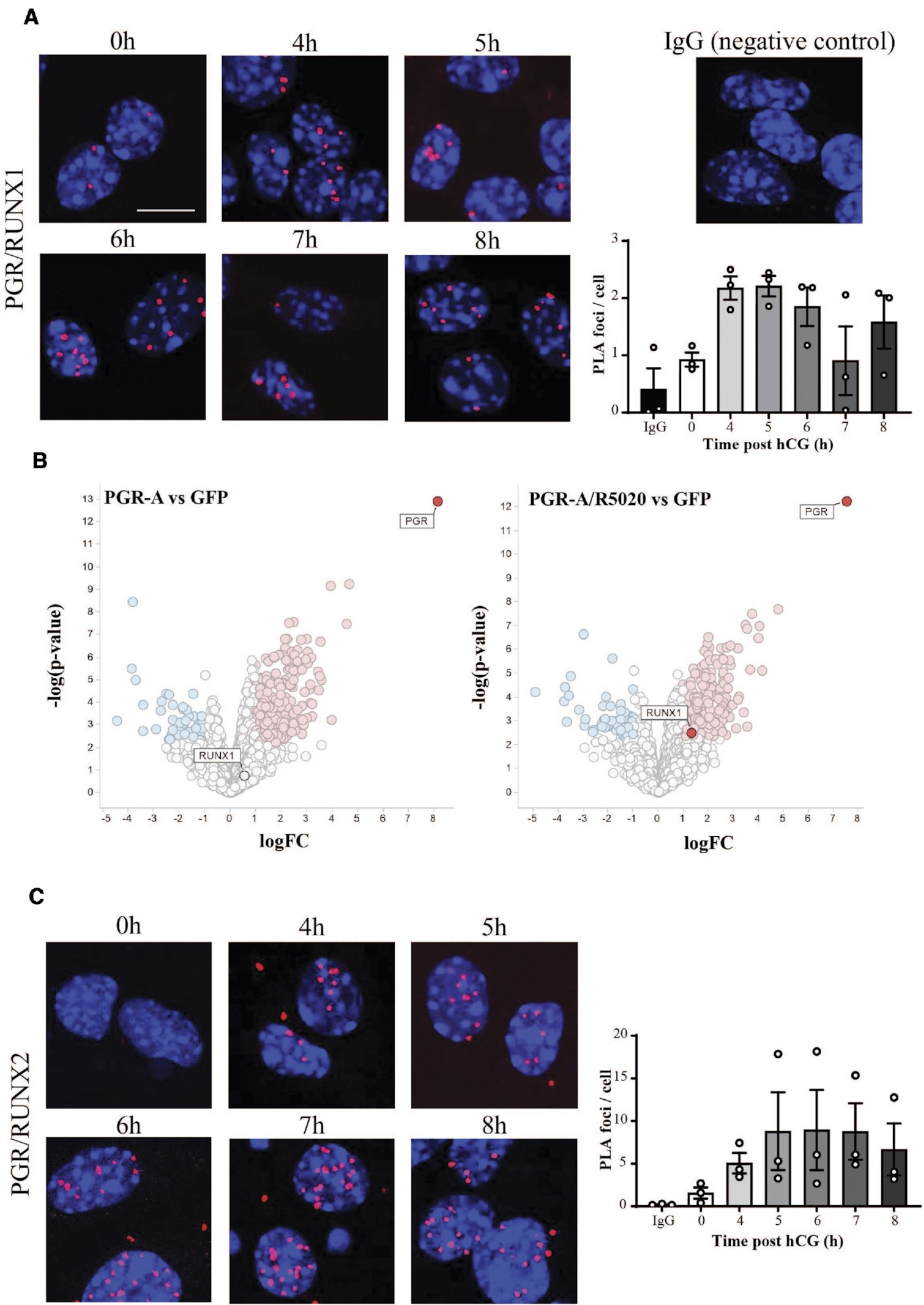
圖4.顆粒細胞中的PGR與RUNX轉錄因子相互作用,以響應黃體生成素激增。
3、PGR介導顆粒細胞染色質可及性
PGR結合區域主要與轉錄活性染色質重疊,如PGR和H3K27ac ChIP-seq峰值重疊75%,以及90%的PGR結合位點與ATAC-seq峰值重疊。此外,在激素刺激的顆粒細胞中還展示了PGR與組蛋白乙酰轉移酶CBP/p300之間的相互作用。通過對PGRWT和PGRKO小鼠顆粒細胞的ATAC-seq進一步研究了PGR結合與染色質可及性之間的功能關系。發現在缺乏PGR的情況下,1499個位點的峰值強度顯著改變(圖5A);有趣的是,在PGRKO中,只有6個位點的ATAC峰值強度增加,而大多數位點在PGRKO中失去了可及性。盡管PGR-染色質結合傾向于顆粒細胞的近端啟動子區域,但PGR依賴的ATAC-seq峰值在整個基因組特征中更均勻地分布(圖5B)。PGR的消融導致了236個基因的失調,通過對PGRWT和PGRKO顆粒細胞的RNA-seq鑒定出這些基因。一致地,在這些PGR依賴基因的啟動子處,ATAC-seq峰值的強度減少是明顯的(Spearman相關系數=0.5116,P值<0.0001)(圖5C)。這表明這些基因的啟動子可及性受到PGR消融的顯著影響。以Gpt2為例,其啟動子在PGRKO中的可及性不同,在PGR缺失時不顯著改變的Abhd2的啟動子可及性也顯示了PGR依賴的染色質可及性(圖5D)。
在PGR驅動的ATAC峰值上進行了轉錄因子模體富集分析,并與PGR ChIP-seq中富集的模體進行了比較。與ChIP-seq一樣,PGR驅動的ATAC峰值序列中最高度富集且最顯著的模體是經典的NR3C模體(相對于背景富集了12.4倍)。與PGR ChIP-seq類似,PGR依賴的ATAC峰值中也顯著富集了一些非經典模體(圖5E)。有趣的是,53%(795個中的1499個)的PGR依賴的ATAC位點被PGR結合,而只有17.8%(267個位點)顯示出任何RUNX1結合,其中幾乎所有位點(250/267個位點)均被PGR和RUNX1結合(圖5F)。此外,PGRKO的ATAC-seq表明PGR通過結合經典NR3C模體直接促進染色質可及性的子集區域。為了驗證這一點,我們使用與PGR密切相關的AR HT-SELEX數據生成了NR3C DNA結合的定量模型。從二核苷酸模型派生的能量logo(15個堿基對)符合NR3C共識或PGR模體。接下來,我們使用該模型估計了每個峰值中的PGR結合親和力,并將其與PGR ChIP-seq峰值強度進行了比較,結果顯示PGR結合得分與PGR結合峰值呈現出適度的線性關系。我們通過受試者工作特征(ROC)曲線分析來檢查DNA結合模型預測PGR染色質占據的能力,發現結合親和力對于所有PGR ChIP位點的染色質占據只有適度的預測能力(AUROC = 0.63,n = 13,976),但在也是PGR依賴的ATAC-seq峰值的PGR ChIP峰值上的預測準確性非常高(AUROC = 0.9,n = 804)(圖5G)。這得到了強烈的支持,因為與所有PGR結合位點相比,PGR綁定的PGR依賴ATAC峰值在ChIP峰值強度和NR3C模體結合親和力上表現出明顯的增強(圖5H)。綜上所述,這表明PGR通過結合選定的染色質區域的NR3C模體來驅動染色質重塑,而在非經典位點上的共享PGR/RUNX結合對于促進染色質可及性的依賴性較小。
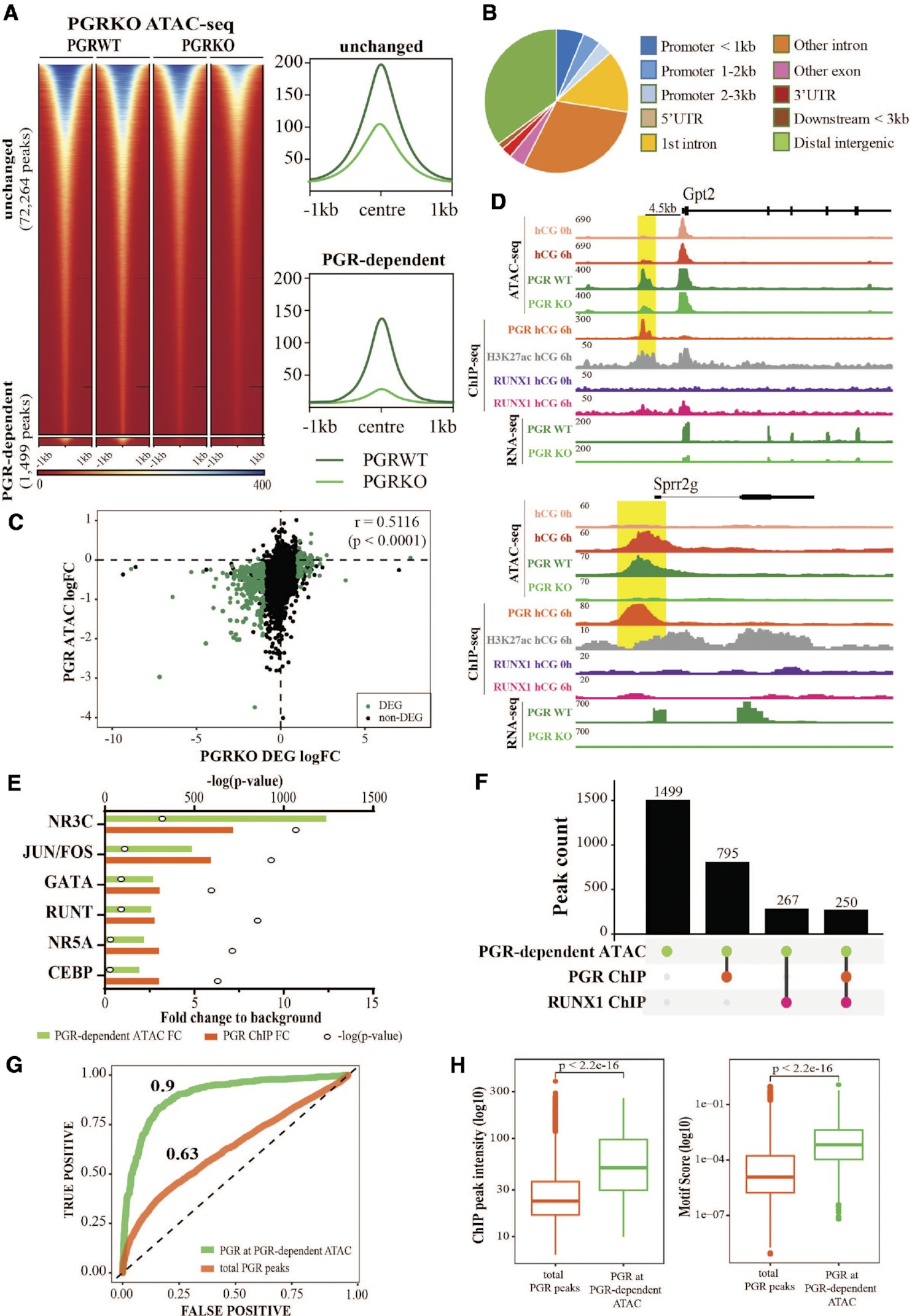
圖5.PGR介導顆粒細胞染色質的可及性。
4、PGR亞型介導特異性排卵基因調控
先前的研究已經在總PGRKO和AKO小鼠中記錄了受干擾的排卵表型,但在BKO小鼠中沒有,通過對這三個小鼠品系的排卵率評估,我們驗證了這一點。我們在從每個品系中獲取的PGRKO、AKO或BKO和WT同胞中的顆粒細胞中進行了RNA-seq,并通過分層聚類發現,AKO顆粒細胞的全局基因表達模式與總PGRKO相似,而BKO則更接近每個品系的WT(圖6A)。在AKO中鑒定了310個基因的差異表達,其中153個(或49.4%)也在缺乏兩個PGR亞型的情況下被鑒定出來(圖6B)。在這些基因中,48.6%和58.5%的AKO和PGRKO差異表達基因分別也在對LH峰值的響應中差異表達。另一方面,失去PGR-B沒有導致顯著的轉錄變化(圖6B)。
為了確定共調控下游PGR靶基因的轉錄因子,使用IPA對DEG數據集的上游調控因子進行了分析(圖6C)。如預期,PGR在PGRKO和AKO數據集中被顯示為最顯著的調控因子。此外,已知被PGR誘導的轉錄因子,如PPARG和HIF1A,也在兩個數據集中富集。重要的是,這種獨立的方法還支持RUNX、JUN/FOS和CBP/p300作為PGR依賴性轉錄反應的介導因子,因為RUNX1、CBFα、JUN、FOS和p300也被通過上游分析鑒定出來。我們在AKO和BKO品系的hCG/孕激素刺激顆粒細胞中進行了PGR/RUNX1和PGR/RUNX2的PLA。在AKO而不是BKO顆粒細胞中觀察到與RUNX1和RUNX2的蛋白質相互作用的減少(圖6D)。
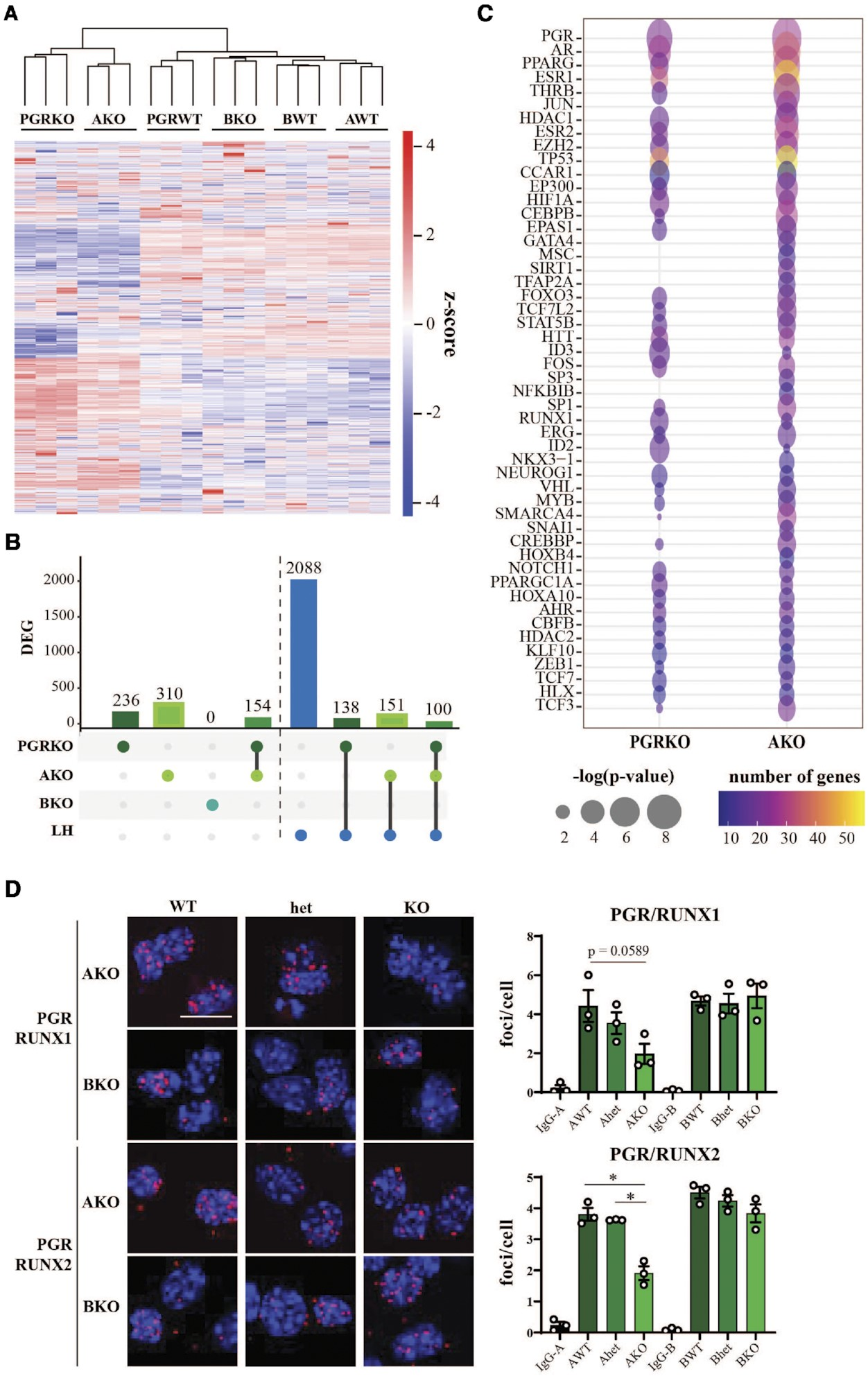
圖6.通過與特定轉錄因子配對的相互作用,圍排卵期顆粒細胞中由PGR亞型驅動的轉錄組。
實驗方法
顆粒細胞培養及激素治療、ATAC-seq、RNA-seq、ChIP-seq、鄰位連接試驗(PLA)、GFP-TRAP免疫沉淀、質譜。
參考文獻
Dinh DT, Breen J, Nicol B, Foot NJ, Bersten DC, Emery A, Smith KM, Wong YY, Barry SC, Yao HHC, Robker RL, Russell DL. Progesterone receptor mediates ovulatory transcription through RUNX transcription factor interactions and chromatin remodelling. Nucleic Acids Res. 2023 Apr 26:gkad271. doi: 10.1093/nar/gkad271. Epub ahead of print. PMID: 37099375.






